Programmziele
Ein Schwerpunktprogramm zun Thema “Robuste Kopplung kontinuumsbiomechanischer in silico Modelle für aktive biologische Systeme als Vorstufe klinischer Applikationen –Co-Design von Modellierung, Numerik und Nutzbarkeit”
Motivation
Das große Potential von in silico (computergestützten) Modellen wird in der Medizin noch wenig genutzt. Wie in anderen hochtechnologischen Bereichen auch, könnte dieses Potential zur beschleunigten Entwicklung von modernen Diagnose- und individualisierten Behandlungsmethoden intensiver genutzt werden. Die meisten existierenden in silico Modelle beschränken sich auf einzelne Skalen (z. B. Zell, Gewebe) oder auf generische Mehrskalenmodelle einzelner Organe (z. B. Muskel, Leber). Um Symptome und Krankheiten zu verstehen, müssen jedoch mehrere Größen- und Zeitskalen im Kontext des gesamten Systems betrachtet werden. Die komplexen skalenübergreifenden Zusammenhänge sind ohne einen systematischen Ansatz und ohne Computermodelle oft nur sehr schwer identifizierbar. Eine zentrale Aufgabe der numerischen Biomechanik und biomedizinischen Forschung ist es daher, robuste Kopplungsmethoden und -strategien zu entwickeln. Diese müssen die Skalen der biologischen Systeme vom Molekül bis zum kompletten Organsystem oder Organismus integrieren.
Herausforderung
Die hohe Komplexität aktiver biologischer Systeme erfordert folglich die enge Zusammenarbeit von Medizin, Ingenieurwissenschaften, Numerischer Mathematik (Numerik) und Informatik. Die Beschreibung mehrskaliger Systemmodelle erfordert insbesondere innovative Kopplungsstrategien, in die modernste Computerarchitekturen, neue und robuste numerische Verfahren sowie Datenstrukturen und -integrationsmöglichkeiten einfließen. Zudem müssen die Simulationsergebnisse in gemeinsamer Arbeit mit Medizinern für den Transfer in die Klinik und für Applikation mit klinischen Fragestellungen aufbereitet werden.
Lösungsansatz
Mit dem beantragten SPP wird ein interdisziplinärer Verbund geschaffen, der sich auf die Erforschung neuer methodischer Ansätze zur Kopplung mehrerer in silico Modelle fokussiert und dessen physiologischen Funktionen und dreidimensionaler Organisation mit berücksichtigt. Gemeinschaftlich werden Fragestellungen definiert und adressiert, die sich übergreifend aus den Bereichen Kopplung (von in silico Modellen), Numerik (robuste und effiziente Algorithmen) und Nutzbarkeit (Standardisierung und Datenintegration, Validierung und Vorbereitung numerischer Kontinuumsmodelle für klinischen Applikationen) zusammensetzen.
Zielsetzung
Ziel des SPPs ist es, die vorhandenen methodischen Grundlagen als Schlüsselqualifikationen weiterzuentwickeln und damit die Generierung robuster biomechanischer Modelle für den Einsatz in der klinischen Praxis zu ermöglichen. Das SPP zielt hingegen nicht darauf ab, den Transfer der Modelle in die Klinik über klinische Studien zu etablieren. Damit versteht sich das SPP als methodisch orientierte Qualifizierung biomechanischer Simulationsmodelle aktiver Systeme für den späteren Einsatz in medizinischen Fragestellungen. Durch das beantragte SPP können die in der deutschen Forschungslandschaft existierenden Kompetenzen nachhaltig und international sichtbar gestärkt werden, wodurch sich eine Vorreiterrolle auf dem Gebiet der kontinuumsbiomechanischen Modellierung von aktiven biologischen Systemen ergibt.
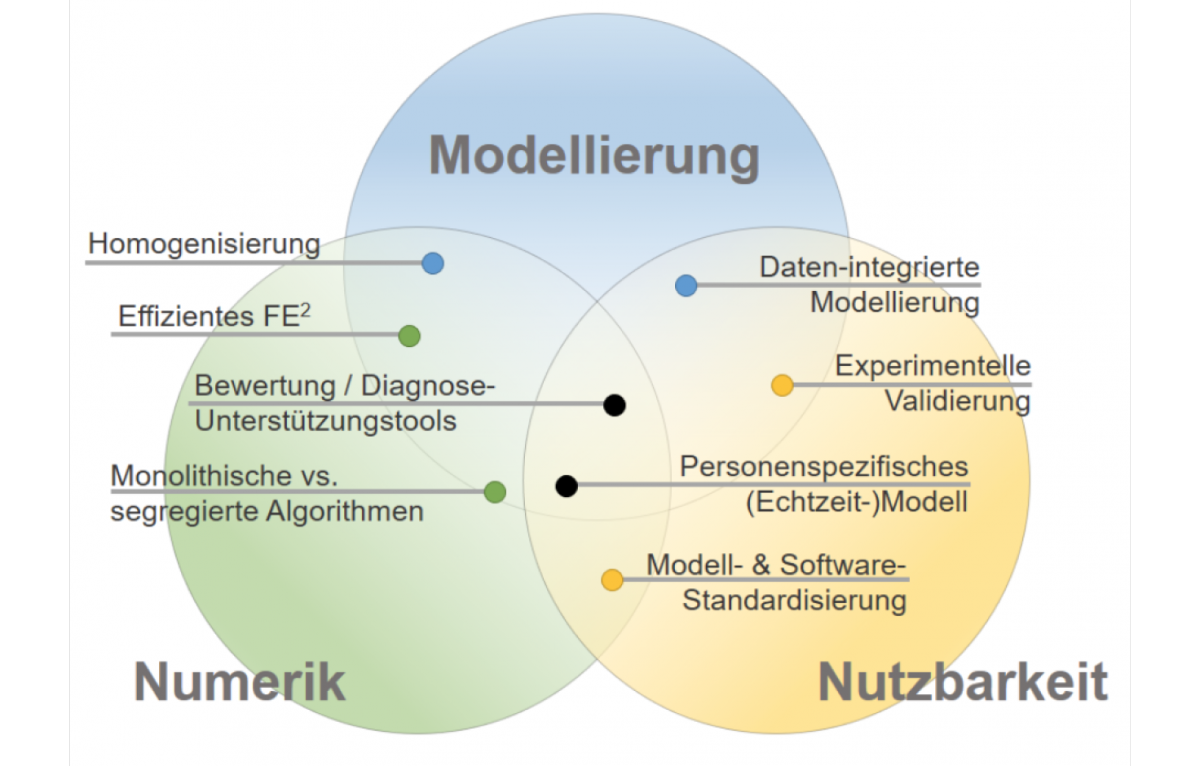
Das SPP ist in drei eng miteinander verzahnte Bereiche untergliedert.
- Der Bereich“Modellierung gekoppelter biomechanischer Systeme” befasst sich auf die Kopplung biomechanischer in silico Modelle zur Beschreibung aktiver biologischer Systeme auf verschiedenen Längen-, Zeit- und/oder Funktionsskalen bzw. auf die Kopplung kontinuumsbiomechanischer Organsystemmodelle.
- Bereich “Numerik gekoppelter biomechanischer Systeme” befasst sich mit mathematische Algorithmen zur robusten, numerischen Lösung gekoppelter Modelle für aktive biologische Systeme sowie deren Implementierung auf Hoch- und Höchstleistungsrechnern.
- Bereich “Nutzbarkeit” befasst sich mit dem Datenaustausch, Validierung und Vorbereitung numerischer kontinuumsbiomechanischer Modelle zur Beantwortung klinisch relevanter Fragestellungen und für personenspezifische Lösungen.
Bereich “Modellierung gekoppelter biomechanischer Systeme”
Biomechanische Modelle werden in vielen Fällen zunächst auf bestimmten Längen- und Zeitskalen entwickelt, die sich an der Funktion oder Struktur orientieren. Wie bereits dargestellt, sind für die Beantwortung klinisch relevanter Fragestellungen skalenübergreifende Modelle und somit die Kopplung unterschiedlicher Skalen notwendig.
Daher liegt auch die zentrale Herausforderung des Bereichs “Modellierung gekoppelter biomechanischer Systeme” in der Modellierung und den mathematischen Beschreibungen von verschiedenen Kopplungsansätzen. Neue Kopplungsstrategieansätze sind beispielsweise für die Überbrückung von Skalensprüngen durch Kopplung detaillierter Modelle benachbarter Skalen notwendig, z.B. durch das Einbinden der Gewebeskala, die meist in mehrskaligen Modellen von Organen ignoriert wird, die Kopplung zweier oder mehrerer funktioneller Einheiten auf der gleichen Skala, z. B. mehrerer Muskeln zu einem muskuloskeletalen System oder die Kopplung zweier dreidimensionaler High-Fidelity-Organmodelle zu einem System.
Modellierungskopplungsmethoden, die innerhalb des SPPs eine Rolle spielen, sind z.B.:
- Diffusion-Advektions-Reaktionsgleichungen (PDE /ODE Kopplung),
- Stoffwechselvorgänge (systembiologische Ansätze, pharmakokinetische Modelle),
- Transport (Blut, Hämodynamik – CFD),
- Homogenisierungsmethoden (Mischungstheorie, Theorie poröser Medien, Biot-Theorie),
- geometrische Kopplung (3D–1D–0D Kopplung) oder
- datengetriebene Kopplung (z.B. Machine Learning als Kopplungswerkzeug).
Beispiele für räumliche und zeitliche Skalen, auf denen die Integration von verschiedenen biophysikalischen Prozessen stattfinden muss, sind
- die zelluläre Ebene (systembiologische Ansätze),
- die Gewebeebene (poröse Strukturen, Mehrkomponentenstrukturen (Fasern, Matrix, Nährstoffe, Ionen), Mehrphasen (Festkörper, Fluid), Perfusionsprozesse),
- die Organebene (mehrskalige, mehrphysikalische Modelle einzelener Organe, z.B. das Herz, ein freipräparierter Muskel, die Leber, Wachstumsprozess, Strömungsprozesse),die Organismusebene (Kopplung Herz und Arterie, mehrerer Muskeln, Leber und Niere) oder
- die Verknüpfung unterschiedlicher Zeitskalen (Kopplung von Wachstums- oder Degenerationsprozessen mit Transportphänomene von Nähr- und Umwandlungsstoffen).
Bereich “Numerik gekoppelter biomechanischer Systeme”
Bei der Erstellung biomechanischer Modelle steht traditionell meist die Modellierung im Vordergrund. Die effiziente Implementierung unter Ausnutzung neuester Algorithmen, Programmierstandards und existierender Softwarebibliotheken wird dabei oft vernachlässigt. Prädiktive numerische Simulationen können jedoch nur erreicht werden, wenn Modellierung und Numerik durch ein starkes Co-Design eng verzahnt sind. Es ist ebenfalls notwendig, um für Modelle hoher Qualität (high-fidelity) schnelle Konvergenz der numerischen Lösungsverfahren zu erreichen.
Eine zentrale Herausforderung für den Bereich “Numerik gekoppelter biomechanischer Systeme” ist die Entwicklung robuster Kopplungsalgorithmen für die verschiedenen gekoppelten Problemklassen, z.B. PDE-PDE, PDE-ODE, ODE-ODE, etc., verschiedene Multiskalenprobleme mit unterschiedlichen räumlichen und / oder zeitlichen Skalen oder Kopplungen über verschiedene Dimensionen, z. B. 0D, 1D, 2D, 3D, etwa um geeignete, nichtreflektierende Randbedingungen zu definieren. Eine weitere Herausforderung ist das Erzielen von paralleler Skalierbarkeit solcher Algorithmen für komplexe Probleme auf modernen Hoch- und Höchstleistungsrechnern. Dazu sollten die Algorithmen direkt so entwickelt werden, dass sie die modernen Rechnerarchitekturen effizient ausnutzen. Da die gekoppelten biomechanischen Modelle in der Regel nichtlinear sein werden, muss die Entwicklung geeigneter numerischer Kopplungsalgorithmen mit aktuellen Algorithmen aus dem Bereich der nichtlinearen Lösungsverfahren kombiniert werden, da sonst häufig keine Konvergenz erzielt werden kann. Hierbei liegt eine große Herausforderung darin, eine Balance zwischen der Stärke der Kopplung (monolithisch vs. segregiert) und der numerischen und parallelen Effizienz, sowie der Robustheit der betrachteten Algorithmen zu erreichen. Für Parameterstudien, Anpassungen an personenspezifisches (Material-) Verhalten und den Transfer zur klinischen Anwendung spielt die numerische Robustheit eine entscheidende Rolle.
Methodische Aspekte, die innerhalb dieses Bereichs eine Rolle spielen, sind
- Kopplungsalgorithmen (monolithisch, segregiert, …),
- Effiziente und parallel skalierende Algorithmen für komplexe mehrskalige Modelle auf Hoch- und Höchstleistungsrechnern,
- Nichtlineare/Lineare Löser (Vorkonditionierung, Gebietszerlegungs- und Mehrgitterverfahren in Ort und Zeit, Kontinuitätsverfahren, nichtlineare Eliminationsverfahren / Gebietszerlegungsverfahren, …), soweit relevant für Kopplungs- und Homogenisierungsalgorithmen, sowie deren Effizienz und Robustheit,
- Modellreduktion (POD, DEIM, neuronale Netze, dünne Gitter, …),
- Numerische Homogenisierungsmethoden (FE2, …) und
- Operator Splitting Methoden.
Bereich “Nutzbarkeit”
Biomechanische in silico Modelle haben das Potential, bestimmte komplexe Vorgänge zu quantifizieren und dadurch z.B. den Nutzen und die detaillierte Planung von chirurgischen Eingriffen vorherzusagen bzw. zu ermöglichen oder deren Umsetzung im Operationssaal zu unterstützen. Weiterhin können sie helfen, Scoring Systeme, wie z. B. das etablierte Bewertungssysteme LiMAx, zu verbessern. Die dabei notwendige Qualitätssicherung der computergestützten Simulationen erfordert die Analyse der Sensitivität einzelner Eingabeparameter auf das Endergebnis im Hinblick auf belastbare Prädiktionen.
Eine der größten Herausforderungen im Bereich “Nutzbarkeit” ist dabei die Erstellung, der Austausch, die Zurverfügungstellung von validierten Modellen und die Einbindung patientenspezifischer Informationen. Dabei spielt neben der Datenintegration auch der Link zwischen ex vivo und in vivo Daten eine zentrale Rolle, um neue Erkenntnisse und eine Verbesserung bestehender Modelle zu erzielen. Im Gegensatz dazu führt die Eigenentwicklung von Software zur Lösung biomechanischer Fragestellungen meist zu Insellösungen, welche den Austausch der Modelle und Daten erschweren und neue Innovationen verhindern.
Durch die Integration von Daten verschiedener Ressourcen und deren intelligente Nutzung, z.B. durch maschinelles Lernen, kann die Zuverlässigkeit und Aussagekraft der in silico Modelle enorm erhöht werden. Zudem müssen Validierungsstudien konzeptioniert werden, damit die Funktionssicherheit komplexer Simulationsmodelle überzeugend nachgewiesen und die Akzeptanz der Anwender erhöht werden kann. Dabei sollte die Robustheit der in silico Modelle im Rahmen klinisch relevanter Sensitivitätsstudien analysiert und experimentell verifiziert werden. Des Weiteren setzt die quantifizierte Analyse mit Hilfe von in silico Modellen voraus, dass entsprechende Tools auch von Medizinern ohne breites technisches Hintergrundwissen anwendbar sind. Die zugrundeliegenden Modelle müssen demzufolge sehr robust und mit einer intuitiven und allgemein verständlichen Benutzeroberfläche ausgestattet sein. Außerdem müssen Methoden gefunden werden, die es erlauben, aus bildgebenden Verfahren morphologische und mechanische Größen zu extrahieren und in robuste dreidimensionale in silico Modelle einfließen zu lassen.
Methodische Aspekte, die innerhalb dieses Bereichs eine Rolle spielen, sind
- Validierung mit
- Co-Design experimenteller Methoden,
- Statistisch gesicherte Validierungsmethoden für gekoppelte biomechanische Systeme,
- Generierung neuer Daten für das Gesamtsystem,
- Simulationsbasierte Analyse und Interpretation inklusive in vivo / ex vivo Link,
- Standardisierung mit
- Datenstandardisierung und Datenverwaltung, z.B. durch Weiterentwicklung von existierenden XML-Standards,
- Integration von Daten verschiedener Ressourcen und deren intelligente Nutzung und
- Anwendung mit
- Datenaufbereitung,
- Echtzeitfähige Systeme, z.B. mit Hilfe von maschinellem Lernen / neuronale Netze,
- Definition und Validierung von Scoring Systemen, z.B. zur Therapie- oder Implantatauswahl durch einen Daten-Simulation-Analyse / Interpretation-Zyklus,
- Erstellung des Workflows in einem klinischen Umfeld, z. B. eine Operationsplanungssoftware, Diagnosesoftware und intuitive Nutzeroberflächen und
- Reflexionsstudien zur Erhöhung der gesellschaftlichen / medizininternen Akzeptanz
- von Simulationsmodellen in der Translation auf klinische Anwendungen.
Thematischen Eingrenzung (Positivliste)
Zur thematischen Eingrenzung sollen in dem SPP nur solche Forschungsaktivitäten gefördert werden, bei denen Kopplungsmethoden eine zentrale Rolle spielen. Des Weiteren sollen explizit nur folgende Themen für die Antragstellung zugelassen werden.
- Für den Bereich Modellierung gekoppelter biomechanischer Systeme sollen sich die im Antrag betrachteten aktiven biomechanischen Systeme mit folgenden Eigenschaften beschäftigen:
- Weichgewebe,
- Mehrphasenmaterialien / Multiphysikalische Modelle,
- Aktive Materialien, z. B. Kraft, Metabolismus oder Signalverarbeitung und
- Kontinuumsmechanische Modelle.
- Für den Bereich Numerik gekoppelter biomechanischer Systeme sollen die Methoden folgende Charakteristika aufweisen:
- Kopplungsalgorithmen zur Lösung gekoppelter Systeme,
- Algorithmen zur Skalenüberbrückung und / oder Homogenisierung, z. B. FE2 und
- Effizienzsteigerung, z. B. Modellreduktion, HPC (Parallelisierung, Datenkommunikation, usw.).
- Für den Bereich Nutzbarkeit sollen folgende Charakteristika berücksichtigt werden:
- Multimodale Daten und Datenintegration,
- Standardisierung, Validität, Qualitätssicherung oder Datenmanagement und
- Relevante medizinische Fragestellungen.
Thematischen Eingrenzung (Negativliste)
Explizit ausgeschlossen werden
- passive biologische Strukturen, wenn nur mechanische Aspekte behandelt werden,
- die Weiterentwicklung von Einskalen- oder Einphasenmodellen,
- rein dynamische Starrkörpermodelle,
- in der ersten Phase des SPPs Projekte zur Unschärfemodellierung,
- Forschungsarbeiten, die sich rein auf subzelluläre Prozesse (-omics, DNA, mRNA, Proteine, Ionenkanäle),
- klinische Studien und
- experimentelle Arbeiten, die nicht im konkreten Zusammenhang mit der Modellierungstehen.
Zwar sind die o.g. Aspekte ebenfalls wichtig für die adressierten Fragestellungen des SPPs, können allerdings auf Grund der nötigen Fokussierung und gewünschten Kohärenz nicht mit aufgenommen werden.
Antragsformate
Anträge sollen in mindestens zwei der drei Bereiche thematisch verankert sein. Angestrebt wird eine Aufteilung der Projekte in
- bis zu 10 Anträge mit 24 Personenmonaten pro Jahr und
- bis zu 5 Anträge mit bis zu 36 Personenmonate pro Jahr.
Beispiele