Ziel:
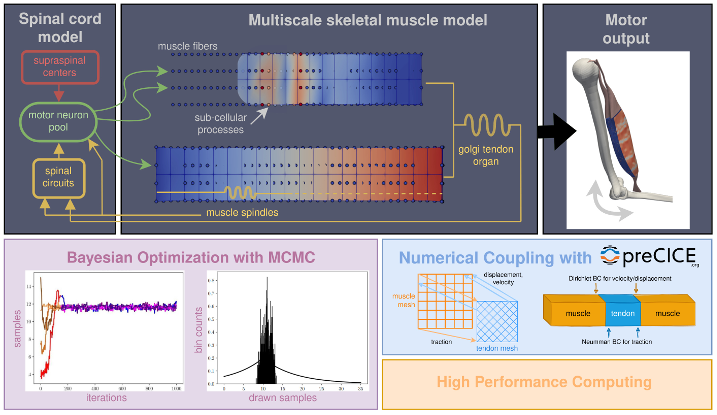
Ziel dieses Projekts ist die Erstellung eines neuartigen In-Silico-Frameworks, das es ermöglicht, Anpassungsreaktionen des neuromuskulären Systems auf Gehirnpathologien zu erklären. Dazu wird durch ein Co-Design zwischen Modell, Mathematik und HPC ein systemisches Mehrskalenmodell des neuromuskulären Systems entwickelt.
Beschreibung:
Ziel dieses Projekts ist die Erstellung eines neuartigen In-Silico-Frameworks, das es ermöglicht, Anpassungsreaktionen auf Gehirnpathologien wie beispielsweise Schlaganfälle, Zerebralparese oder multiple Sklerose im neuromuskulären System zu erklären. Solche Pathologien schränken die motorischen Fähigkeiten der Betroffenen erheblich ein, zufriedenstellende Behandlungsmöglichkeiten existieren allerdings nicht.
Letztendlich ist es das Ziel, die Entwicklung neuartiger und die Verbesserung existierender therapeutischer Maßnahmen zu unterstützen. Basierend auf der Idee, dass der Körper versucht, sich so anzupassen, dass die gegebenen Bedingungen optimal gehandhabt werden, wollen wir mathematische Methoden zur Optimierung unter Nebenbedingungen zur Erreichung des Projektziels einsetzen. Indem wir ein systemisches Mehrskalenmodell des neoromuskulären Systems verwenden, erwarten wir aussagekräftige Vorhersagen für reale physiologische Systeme. Angesichts der hohen Komplexität, die ein solches Framework für die Modellierung, Rechenlast und Mathematik bedeutet, wurde ein solcher Ansatz bisher nicht verfolgt. Zur Zielerreichung wollen wir unser neuromuskuläres Mehrskalenmodell mit unserem 3D-Kontinuumsmechanikmodell vereinen.
Dafür sind folgende Beiträge geplant: Wir werden neue mathematische Modelle für Motorkontrolle und Gehirnschädigungen integrieren und die existierenden neuromuskulären Modelle um heteronyme Feedback-Schleifen, Remodellierungsprozesse und Muskelmetabolismus erweitern. Um eine flexible Simulations- und Optimierungsumgebung mit austauschbaren Komponenten zu erhalten, streben wir einen partitionierten Ansatz an. Dies erfordert neue technische und numerische Kopplungsmethoden sowie Konzepte zur Handhabung der Mehrskaligkeit zwischen kurz- und langfristigen Reaktionen auf Gehirnpathologien. Die Optimierung basierend auf diesen neuen Modellen erfordert wiederum Co-Design zwischen Modell, Mathematik und HPC: (i) Zielfunktionen, die High-level-Ziele des neuromuskulären Systems wiedergeben sowie Optimierungsparameter, d.h. die Freiheitsgrade des neuromukulären Systems, die zulässige kurz- und langfristige Anpassungen auf gegebene Störungen repräsentieren, (ii) weitere Komponenten des Optimierungsframeworks müssen entwickelt werden, insbesondere Surrogate zur Reduktion der Rechenlast, adjungierte Löser im Langrangeansatz, und der äußere Optimierer selbst.
Für mögliche klinische Anwendungen (jenseits der Möglichkeiten dieses Projekts) müssen weitere Herausforderungen im Hinblick auf Datenhandhabung in unserem modularen Optimierungsframework berücksichtigt werden. Alle Ziele erfordern eine enge Zusammenarbeit zwischen den Experten in den Gruppen der PIs im Sinne des Co-Designs zwischen Modellen, Numerik, HPC und Daten.
Involvierte Institutionen:
Universität Stuttgart
Institut für Angewandte Analysis und Numerische Simulation
Mathematische Methoden für komplexe Simulation der Naturwissenschaft und Technik
Universität Stuttgart
Institut für Parallele und Verteilte Systeme
Abteilung Simulation großer Systeme
Universität Stuttgart
Institut für Modellierung und Simulation Biomechanischer Systeme
Kontinuumsbiomechanik und Mechanobiologie
Links:
Antragsteller:
Prof. Dr. rer. nat. Dominik Göddeke, Universität Stuttgart, Institut für Angewandte Analysis u. Numerische Simulation, Mathematische Methoden für komplexe Simulation der Naturwissenschaft und Technik
Prof. Dr. rer. nat. habil. Miriam Schulte, Universität Stuttgart, Institut für Parallele und Verteilte Systeme, Abteilung Simulation großer Systeme
Prof. Oliver Röhrle, PhD, Universität Stuttgart, Institut für Modellierung und Simulation Biomechanischer Systeme, Kontinuumsbiomechanik und Mechanobiologie
Publikationen:
2024
OpenDiHu: An efficient and scalable framework for biophysical simulations of the neuromuscular system Journal Article
In: Journal of Computational Science, iss. 79, pp. 102291, 2024, ISBN: 1877-7503.
Coupled simulations and parameter inversion for neural system and electrophysiological muscle models Journal Article
In: GAMM-Mitteilungen, 2024.
Postinhibitory excitation in motoneurons Journal Article
In: PLOS Computational Biology, vol. 20, iss. 1, 2024.
2023
Linking cortex and contraction—Integrating models along the corticomuscular pathway Journal Article
In: Frontiers in Physiology, Sec. Computational Physiology and Medicine , iss. Vol 14-2023, 2023.
2022
preCICE v2: A sustainable and user-friendly coupling library Journal Article
In: Open Research Europe, 2022.
Spindle Model Responsive to Mixed Fusimotor Inputs: an updated version of the Maltenfort and Burke (2003) model Journal Article
In: Physiome, 2022.
2020
POD-DEIM Model Order Reduction for the Monodomain Reaction-Diffusion Sub-Model of the Neuro-Muscular System Journal Article
In: IUTAM Symposium on Model Order Reduction of Coupled Systems, Stuttgart, Germany, May 22–25, 2018, pp. 177–190, 2020.
